réservé à la recherche
UNC1999 Histone Methyltransferase inhibiteur
N° Cat.S7165
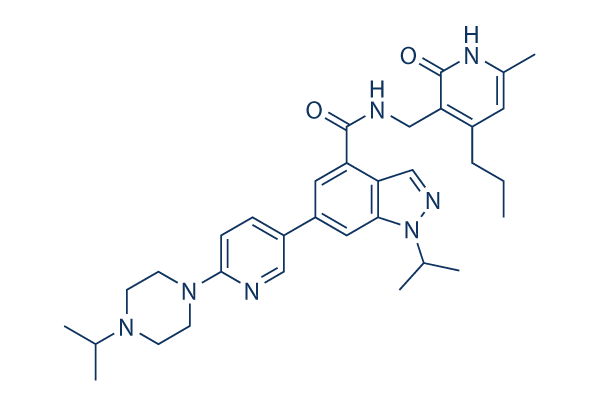
Structure chimique
Poids moléculaire: 569.74
Contrôle qualité
| Cibles apparentées | HDAC JAK BET PKC PARP HIF PRMT EZH2 AMPK Histone Acetyltransferase |
|---|---|
| Autre Histone Methyltransferase Inhibiteurs | Pinometostat (EPZ5676) 3-Deazaneplanocin A (DZNep) Hydrochloride BIX-01294 trihydrochloride EPZ015666 (GSK3235025) EPZ004777 MM-102 (HMTase Inhibitor IX) Chaetocin SGC 0946 EPZ005687 UNC0638 |
Culture cellulaire, traitement et concentration de travail
| Lignées cellulaires | Type dessai | Concentration | Temps dincubation | Formulation | Description de lactivité | PMID |
|---|---|---|---|---|---|---|
| human MCF10A cells | Function assay | 72 h | Inhibition of EZH2 in human MCF10A cells assessed as reduction of H3K27me3 level after 72 hrs by Western blot analysis, IC50=0.124 μM | 25406853 | ||
| human MCF10A cells | Cytotoxic assay | Cytotoxicity against human MCF10A cells assessed as cell viability by Alamar Blue assay, EC50=19.2 μM | 25406853 | |||
| Cliquez pour voir plus de données expérimentales sur les lignées cellulaires | ||||||
Informations chimiques, stockage et stabilité
| Poids moléculaire | 569.74 | Formule | C33H43N7O2 |
Stockage (À partir de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 1431612-23-5 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | N/A | Smiles | CCCC1=C(C(=O)NC(=C1)C)CNC(=O)C2=C3C=NN(C3=CC(=C2)C4=CN=C(C=C4)N5CCN(CC5)C(C)C)C(C)C | ||
Solubilité
|
In vitro |
DMSO
: 100 mg/mL
(175.51 mM)
Ethanol : 100 mg/mL Water : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Entrez les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Entrez la formulation in vivo (Ceci nest que le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuiteμL PEG300, mélanger et clarifier, ajouter ensuiteμL Tween 80, mélanger et clarifier, ajouter ensuite μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuite μL Huile de maïs, mélanger et clarifier.
Remarque : 1. Assurez-vous que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue lors de lajout précédent est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Fonctionnalités |
The first orally bioavailable inhibitor against wild-type and mutant EZH2 as well as EZH1.
|
|---|---|
| Targets/IC50/Ki |
EZH2
(Cell-free assay) 2 nM
EZH1
(Cell-free assay) 45 nM
|
| In vitro |
L'UNC1999 est très puissant pour les mutants EZH2 Y641N et EZH2 Y641F in vitro. Ce composé provoque des réductions concentration-dépendantes de H3K27me3 dans les cellules MCF10A avec un IC50 de 124 nM, tout en montrant une faible toxicité cellulaire. Il présente une inhibition puissante et concentration-dépendante de la prolifération cellulaire avec une EC50 de 633 nM dans une lignée cellulaire de DLBCL hébergeant le mutant EZH2Y641N. De plus, l'UNC1999 biotinylé enrichit EZH2 à partir de lysats de cellules HEK293T, et peut donc être utilisé dans des études de chimioprotéomique.
|
| Kinase Assay |
Test de proximité par scintillation
|
|
Les tests d'activité de la méthyltransférase sont effectués en surveillant l'incorporation d'un groupe méthyle tritié provenant de la S-adénosylméthionine (3H-SAM) dans des substrats peptidiques biotinylés à l'aide du Scintillation Proximity Assay (SPA) pour le complexe trimérique PRC2-EZH2 (EZH2:EED:SUZ12), le complexe pentamérique PRC2-EZH1 (EZH1:EED:SUZ12:RBBP4:AEBP2), SETD7, G9a, GLP, SETDB1, SETD8, SUV420H1, SUV420H2, SUV39H2, le complexe tétramérique MLL1 (MLL:WDR5:RbBP5:ASH2L), PRMT1, PRMT3, le complexe PRMT5-MEP50 et SMYD2. Le tampon de réaction pour SMYD2 et SMYD3 est 50 mM Tris pH 9.0, 5 mM DTT, 0.01% TritonX-100 ; pour G9a, GLP et SUV39H2 est 25 mM phosphate de potassium pH 8.0, 1 mM EDTA, 2 mM MgCl2 et 0.01% Triton X-100 ; et pour les autres HMT 20 mM Tris pH 8.0, 5 mM DTT, 0.01% TritonX-100. Pour arrêter les réactions enzymatiques, 10 μL de 7.5 M chlorhydrate de guanidine sont ajoutés, suivis de 180 μL de tampon, mélangés et transférés dans une FlashPlate à 384 puits. Après mélange, les mélanges réactionnels sont incubés et les comptes CPM sont mesurés à l'aide d'un lecteur de plaques Topcount. Les comptes CPM en l'absence de ce composé pour chaque ensemble de données sont définis comme 100% d'activité. En l'absence de l'enzyme, les comptes CPM dans chaque ensemble de données sont définis comme le bruit de fond (0%). Les valeurs d'IC50 sont déterminées en utilisant des concentrations de composé allant de 100 nM à 100 μM. Les valeurs d'IC50 sont déterminées à l'aide du logiciel SigmaPlot. Les essais EZH2-Y641F sont effectués en utilisant 30 nM d'enzyme dans 20 mM Tris pH 8, 5 mM DTT, 0.01% Triton X-100, 5 μM SAM et 1 μM de peptide H3 (1-24) (identique à celui du complexe PRC2-EZH2 de type sauvage). Pour DNMT1, l'essai est effectué en utilisant de l'ADN double brin hémi-méthylé comme substrat. Le substrat d'ADN double brin est préparé par recuit de deux brins complémentaires (brin sens biotinylé : BGAGCCCGTAAGCCCGTTCAGGTCG et brin antisens : CGACCTGAACGGGCTTACGGGCTC), synthétisés par Eurofins MWG Operon. Le tampon de réaction est 20 mM Tris-HCl, pH 8.0, 5 mM DTT, 0.01% Triton X-100. Les essais d'activité méthyltransférase pour DOT1L sont effectués à l'aide de plaques filtrantes. Les mélanges réactionnels dans 20 mM Tris-HCl, pH 8.0, 5 mM DTT, 2 mM MgCl2 et 0.01% Triton X-100 sont incubés à température ambiante pendant 1h, 100 μL de TCA à 10% sont ajoutés, mélangés et transférés sur une plaque filtrante. Les plaques sont centrifugées à 2000 tr/min pendant 2 min, suivies de 2 lavages supplémentaires avec du TCA à 10% et un lavage à l'éthanol (180 μL) suivi d'une centrifugation. Les plaques sont séchées et 100 μL de MicroO sont ajoutés et centrifugés. 70 μL de MicroO sont ajoutés et les CPM sont mesurés à l'aide d'un lecteur de plaques Topcount.
|
|
| In vivo |
Le traitement par UNC1999 (150 et 50 mg/kg, i.p.) entraîne des concentrations plasmatiques de ce composé supérieures à son IC50 cellulaire sur 24 heures in vivo. De plus, il est également biodisponible par voie orale chez la souris, ce qui rend les études animales chroniques plus pratiques et commodes.
|
Références |
|
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
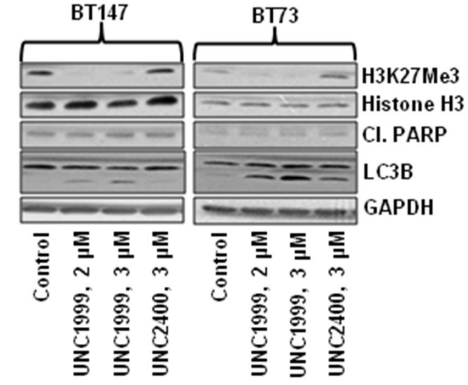
| Western blot | H3K27Me3 / Cleaved PARP / LC3B H3K27me2 / H3K27me1 / H3K27ac / H3K4me3 / H3K9me2 / H3K36me2 |
|
27449082 |
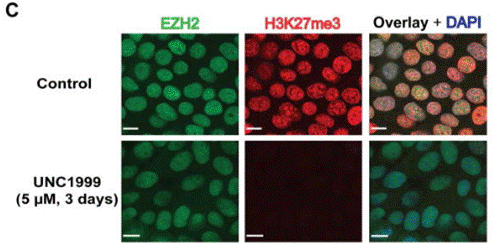
| Immunofluorescence | EZH2 / H3K27me3 NF-κB |
|
23614352 |
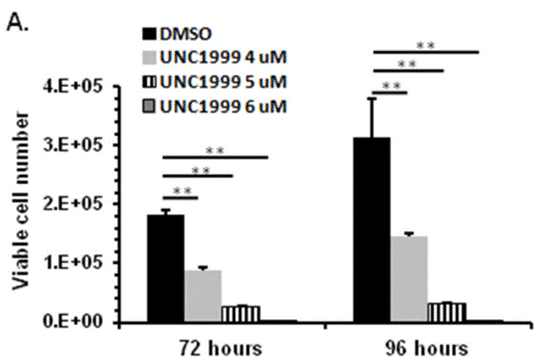
| Growth inhibition assay | Cell viability |
|
27449082 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
