réservé à la recherche
Selisistat (EX-527) inhibiteur de SIRT1
N° Cat.S1541
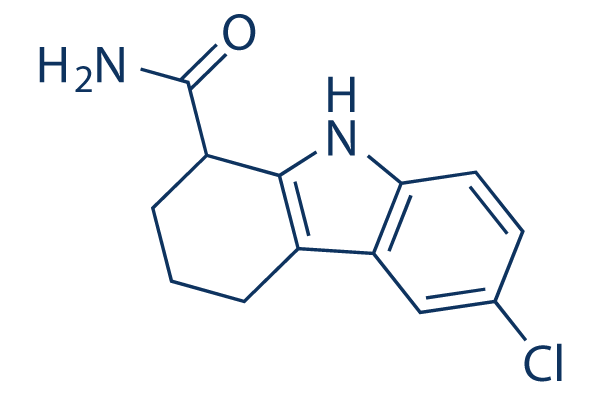
Structure chimique
Poids moléculaire: 248.71
Contrôle qualité
| Cibles apparentées | HDAC JAK BET Histone Methyltransferase PKC PARP HIF PRMT EZH2 AMPK |
|---|---|
| Autre Sirtuin Inhibiteurs | SRT1720 HCl Sirtinol Fisetin 3-TYP AGK2 SRT2104 (GSK2245840) OSS_128167 SirReal2 Thiomyristoyl NRD167 |
Culture cellulaire, traitement et concentration de travail
| Lignées cellulaires | Type dessai | Concentration | Temps dincubation | Formulation | Description de lactivité | PMID |
|---|---|---|---|---|---|---|
| Platelets | Apoptosis Assay | 10/50 μM | 10 min | DMSO | increases ROS level in a dose-dependent manner | 25829495 |
| HEK 293 | Growth Inhibition Assay | 24 h | IC50=97.7 ± 8.1 μM | 24998427 | ||
| HeLa | Growth Inhibition Assay | 24 h | IC50=37.9 ± 1.8 μM | 24998427 | ||
| HEK 293 | Growth Inhibition Assay | 48 h | IC50=69.0 ± 0.7 μM | 24998427 | ||
| HeLa | Growth Inhibition Assay | 48 h | IC50=8.9 ± 1.9 μM | 24998427 | ||
| RMECs | Apoptosis Assay | 10 μM | 24 h | attenuates the anti-apoptotic effect of Des-G | 24486147 | |
| HUVECs | Apoptosis Assay | 10 μM | 24 h | bolishes the protective effect of resveratrol in cell viability | 23358928 | |
| K562 | Function Assay | 0.1-1 μM | 2 h | stimulates Nrf2-dependent gene transcription | 21196497 | |
| INS-1E | Function Assay | 1 μm | 24 h | prevents resveratrol-induced up-regulation of Glut2, glucokinase,Pdx-1, and Tfam | 21163946 | |
| MCF-7 | Growth Inhibition Assay | 0-100 μM | 24/48/72 h | DMSO | represses cell proliferation at the concentration ≥100 μM | 20371709 |
| NCI-H460 | Function Assay | 1 μm | 6 h | DMSO | produces a concentration-dependent increase in the amount of acetylated p53 | 16354677 |
| 293T | Function assay | Inhibition of human recombinant GST-tagged SIRT1 expressed in 293T cells by Fluor de Lys fluorescence assay, IC50 = 0.038 μM. | 21306906 | |||
| Escherichia coli cells | Function assay | Inhibition of human recombinant SIRT1 expressed in Escherichia coli cells using acetylated Lys side chain amino acids 379-382 (Arg-His-Lys-Lys(Ac)) p53 conjugated with aminomethylcoumarin as substrate by fluorescence assay, IC50 = 0.16 μM. | 22931526 | |||
| BL21 (DE3) | Function assay | Inhibition of full length human SIRT1 expressed in Escherichia coli BL21 (DE3) cells using fluorogenic 7-amino-4-methylcoumarin (AMC)-labeled peptide by fluorescence assay, IC50 = 0.21 μM. | 25275824 | |||
| Namalwa cells | Function assay | 3 hrs | Inhibition of SIRT1-mediated endogenous p53 deacetylase activity in human Namalwa cells assessed as concentration required to enhance p53 deacetylation to twice the basal level after 3 hrs by ELISA, INH = 0.6 μM. | 25971769 | ||
| BL21 (DE3) | Function assay | Inhibition of full length human SIRT2 expressed in Escherichia coli BL21 (DE3) cells using fluorogenic 7-amino-4-methylcoumarin (AMC)-labeled peptide by fluorescence assay, IC50 = 1.94 μM. | 25275824 | |||
| Escherichia coli cells | Function assay | Inhibition of human recombinant SIRT2 expressed in Escherichia coli cells using acetylated Lys side chain amino acids 379-382 (Arg-His-Lys-Lys(Ac)) p53 conjugated with aminomethylcoumarin as substrate by fluorescence assay, IC50 = 48.5 μM. | 22931526 | |||
| A673 | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for A673 cells | 29435139 | |||
| SK-N-MC | qHTS assay | qHTS of pediatric cancer cell lines to identify multiple opportunities for drug repurposing: Primary screen for SK-N-MC cells | 29435139 | |||
| Hs683 | Cell cycle assay | 24 to 48 hrs | Cell cycle arrest in human Hs683 cells assessed as accumulation at G1 phase at IC50 after 24 to 48 hrs by propidium iodide staining-based flow cytometry | 28475330 | ||
| HCT116 | Function assay | 10 uM | 8 hrs | Inhibition of SIRT1 in human HCT116 cells assessed as increase in acetylated p53 at 10 uM after 8 hrs by Western blot analysis | 22642300 | |
| NCI-H460 | Function assay | 6 hrs | Inhibition of SIRT-2 in human NCI-H460 cells assessed as inhibition of p53 deacetylation after 6 hrs by immunoprecipitation/immunoblotting analysis, IC50 = 1 μM. | ChEMBL | ||
| Cliquez pour voir plus de données expérimentales sur les lignées cellulaires | ||||||
Informations chimiques, stockage et stabilité
| Poids moléculaire | 248.71 | Formule | C13H13ClN2O |
Stockage (À partir de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 49843-98-3 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | SEN0014196 | Smiles | C1CC(C2=C(C1)C3=C(N2)C=CC(=C3)Cl)C(=O)N | ||
Solubilité
|
In vitro |
DMSO
: 300 mg/mL
(1206.22 mM)
Ethanol : 30 mg/mL Water : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Entrez les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Entrez la formulation in vivo (Ceci nest que le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuiteμL PEG300, mélanger et clarifier, ajouter ensuiteμL Tween 80, mélanger et clarifier, ajouter ensuite μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuite μL Huile de maïs, mélanger et clarifier.
Remarque : 1. Assurez-vous que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue lors de lajout précédent est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Fonctionnalités |
Greater potency, specificity, stability, and lower toxicity than other inhibitors of SIRT1 catalytic activity identified to date.
|
|---|---|
| Targets/IC50/Ki |
SIRT1
(Cell-free assay) 38 nM
|
| In vitro |
Selisistat (EX 527) exerce un effet inhibiteur puissant contre l'activité déacétylase de SIRT1 de manière concentration-dépendante avec une IC50 de 38 nM, et présente une activité beaucoup plus faible contre SIRT2 et SIRT3 avec des valeurs d'IC50 de 19,6 µM et 48,7 µM, respectivement. Il n'inhibe pas l'activité des SIRT4-7 et des HDAC de classe I/II à des concentrations allant jusqu'à 100 µM. Ce composé seul (1 µM) n'a pas d'effet détectable sur l'acétylation de la lysine 382 de p53 dans les cellules NCI-H460, mais augmente significativement la quantité de p53 acétylée dans les cellules NCI-H460, les cellules épithéliales mammaires humaines, les cellules U-2 OS et MCF-7 soumises à des agents génotoxiques comme le peroxyde d'hydrogène, ce qui est plus efficace que celui causé par le Nicotinamide (5 mM). Étonnamment, il n'entraîne pas d'effets détectables sur l'expression génique contrôlée par p53, la survie cellulaire ou la prolifération cellulaire. Il provoque une augmentation de 90% du nombre de cellules HCT116 après 7 jours dans la condition de 0,1% de sérum mais pas de 10% de sérum, suggérant que SIRT1 est un régulateur significatif de la prolifération cellulaire pendant les conditions de privation de facteur de croissance. De plus, il annule les effets du resvératrol sur les réponses au glucose et prévient la régulation positive induite par le resvératrol de Glut2, glucokinase, Pdx-1 et Tfam dans les cellules INS-1E, en raison de l'effet opposé d'EX 527 et du resvératrol sur l'activité déacétylase de SIRT1. |
| Kinase Assay |
Inhibition de l'activité déacétylase de GST-SIRT1
|
|
Les cellules 293T sont transfectées transitoirement avec SIRT1 humain marqué GST dans le vecteur pDEST27 Gateway à l'aide de FuGENE-6. Après 48 heures, les cellules sont lysées avec 50 mM Tris, pH 8,0, 120 mM NaCl, 1 mM EDTA et 0,5% de Nonidet P-40, complétés par des comprimés de cocktail d'inhibiteurs de protéase Complete Mini. La GST-SIRT1 est purifiée à partir des lysats et lavée abondamment dans le tampon ci-dessus. Le test de déacétylation est réalisé avec environ 30 ng de GST-SIRT1 en présence de Selisistat (EX 527) (48 pM à 100 μM). La déacétylation est mesurée à l'aide du kit Fluor de Lys en utilisant un peptide fluorogénique comprenant les résidus 379 à 382 de p53, acétylé sur la lysine 382. Le résidu de lysine acétylé est couplé à une fraction aminométhylcoumarine. Le peptide est déacétylé par SIRT1, suivi de l'ajout d'un développeur protéolytique qui libère l'aminométhylcoumarine fluorescente. En bref, les préparations enzymatiques sont incubées avec 170 μM de NAD+ et 100 μM de peptide fluorogénique p53 pendant 45 minutes à 37 °C, suivies d'une incubation dans le développeur pendant 15 minutes à 37 °C. La fluorescence est mesurée par excitation à 360 nm et émission à 460 nm et l'activité enzymatique est exprimée en unités de fluorescence relative.
|
|
| In vivo |
L'administration de Selisistat (EX 527 ; ~10 µg) à des rats augmente les niveaux d'acétyl-p53 hypothalamiques en inhibant l'activité de SIRT1 hypothalamique. La co-administration de ce composé avec la ghréline atténue fortement l'action orexigène de la ghréline en diminuant les niveaux de pAMPK, en augmentant les niveaux d'ACC et en abolissant l'expression plus élevée des facteurs de transcription FoxO1, pCREB et Bsx et des neuropeptides NPY et AgRP dans le noyau arqué hypothalamique. |
Références |
|
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
| Western blot | GRP78 / FASL / Bcl-2 / LC3 Ac-H3K9 / Fibronectin / Collagen 1 / α-SMA PCNA / Cyclin D1 / Cyclin E pEGFR / EGFR / pPDGFRβ / PDGFRβ pSTAT3 / STAT3 p27 FOXO3a / SIRT1 / Ac-p53 / p16(INK4a) |
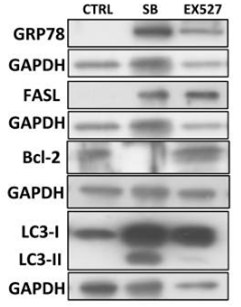
|
29312612 |
| Immunofluorescence | p-p38 |
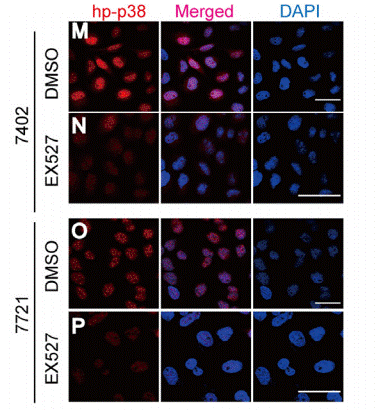
|
26824501 |
| Growth inhibition assay | Cell viability |
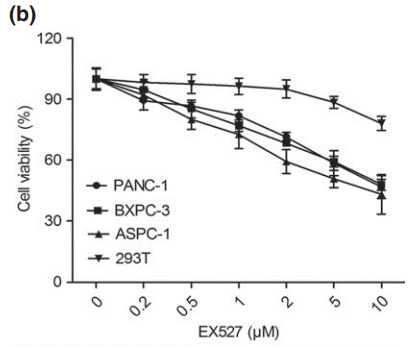
|
24484175 |
Informations sur lessai clinique
(données de https://clinicaltrials.gov, mis à jour le 2024-05-22)
| Numéro NCT | Recrutement | Conditions | Sponsor/Collaborateurs | Date de début | Phases |
|---|---|---|---|---|---|
| NCT01485965 | Completed | Huntington''s Disease |
Siena Biotech S.p.A. |
November 2011 | Phase 1 |
| NCT01521832 | Completed | Huntington''s Disease |
Siena Biotech S.p.A. |
October 2009 | Phase 1 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
Questions fréquemment posées
Question 1:
what is the extinction coefficient of it?
Réponse :
Its extinction coefficient is 1421.650635.
