réservé à la recherche
P5091 DUB inhibiteur
N° Cat.S7132
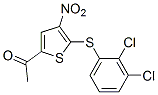
Structure chimique
Poids moléculaire: 348.22
Contrôle qualité
| Cibles apparentées | Proteasome E1 Activating E3 Ligase p97 SUMO E2 conjugating |
|---|---|
| Autre DUB Inhibiteurs | PR-619 IU1 P22077 b-AP15 ML323 LDN-57444 VLX1570 TCID EOAI3402143 ML364 |
Culture cellulaire, traitement et concentration de travail
| Lignées cellulaires | Type dessai | Concentration | Temps dincubation | Formulation | Description de lactivité | PMID |
|---|---|---|---|---|---|---|
| Sf9 cells | Function assay | Inhibition of recombinant USP7 expressed in Sf9 cells by Ub-CHOP reporter assay, EC50=4.2 μM | 24900381 | |||
| human HCT116 cells | Cytotoxic assay | 72 h | Cytotoxicity against human HCT116 cells after 72 hrs, EC50=11 μM | 24900381 | ||
| multiple myeloma cells | Function assay | Inhibition of USP7 in multiple myeloma cells (unknown origin) by immunohistochemistry, EC50 = 4.2 μM. | 25364867 | |||
| Sf9 | Function assay | 30 mins | Inhibition of full-length recombinant USP7 (unknown origin) expressed in Sf9 cells using Ub-EKL as substrate preincubated for 30 mins followed by substrate addition by fluorescence based assay, IC50 = 4.2 μM. | 28768102 | ||
| Cliquez pour voir plus de données expérimentales sur les lignées cellulaires | ||||||
Informations chimiques, stockage et stabilité
| Poids moléculaire | 348.22 | Formule | C12H7Cl2NO3S2 |
Stockage (À partir de la date de réception) | |
|---|---|---|---|---|---|
| N° CAS | 882257-11-6 | Télécharger le SDF | Stockage des solutions mères |
|
|
| Synonymes | P005091 | Smiles | CC(=O)C1=CC(=C(S1)SC2=C(C(=CC=C2)Cl)Cl)[N+](=O)[O-] | ||
Solubilité
|
In vitro |
DMSO
: 28 mg/mL
(80.4 mM)
Water : Insoluble Ethanol : Insoluble |
Calculateur de molarité
|
In vivo |
|||||
Calculateur de formulation in vivo (Solution claire)
Étape 1 : Entrez les informations ci-dessous (Recommandé : Un animal supplémentaire pour tenir compte des pertes pendant lexpérience)
Étape 2 : Entrez la formulation in vivo (Ceci nest que le calculateur, pas la formulation. Veuillez nous contacter dabord sil ny a pas de formulation in vivo dans la section Solubilité.)
Résultats du calcul :
Concentration de travail : mg/ml;
Méthode de préparation du liquide maître DMSO : mg médicament prédissous dans μL DMSO ( Concentration du liquide maître mg/mL, Veuillez nous contacter dabord si la concentration dépasse la solubilité du DMSO du lot de médicament. )
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuiteμL PEG300, mélanger et clarifier, ajouter ensuiteμL Tween 80, mélanger et clarifier, ajouter ensuite μL ddH2O, mélanger et clarifier.
Méthode de préparation de la formulation in vivo : Prendre μL DMSO liquide maître, ajouter ensuite μL Huile de maïs, mélanger et clarifier.
Remarque : 1. Assurez-vous que le liquide est clair avant dajouter le solvant suivant.
2. Assurez-vous dajouter le(s) solvant(s) dans lordre. Vous devez vous assurer que la solution obtenue lors de lajout précédent est une solution claire avant de procéder à lajout du solvant suivant. Des méthodes physiques telles que le vortex, les ultrasons ou le bain-marie peuvent être utilisées pour faciliter la dissolution.
Mécanisme daction
| Targets/IC50/Ki |
USP7
(Cell-free assay) 4.2 μM(EC50)
USP47
(Cell-free assay) 4.3 μM
|
|---|---|
| In vitro |
P5091 est un thiophène trisubstitué avec des substituants dichlorophénylthio, nitro et acétyle médiant l'activité anti-USP7. Ce composé présente une activité déubiquitinylante puissante, spécifique et sélective contre USP7. En revanche, il n'inhibe pas d'autres DUBs ou d'autres familles de protéases à cystéine testées (EC50 > 100 mM). Ce produit chimique inhibe le marquage de l'USP7 avec HA-UbVME de manière concentration-dépendante. Le clivage médiatisé par USP7 des chaînes de polyubiquitine de haut poids moléculaire est inhibé de manière dose-dépendante par ce composé. De plus, il inhibe le clivage des chaînes de Ubiquitin poly-liées en K48 médiatisé par USP7, mais pas par USP2 ou USP8. L'inhibition de l'USP7 par ce composé induit la polyubiquitinylation de HDM2 et accélère la dégradation de HDM2. Il inhibe l'activité déubiquitinylante de l'USP7, sans bloquer l'activité du protéasome dans les cellules MM. Ce produit chimique induit une diminution dose-dépendante de la viabilité de diverses lignées cellulaires MM. Il surmonte la croissance des cellules MM induite par les cellules stromales de la moelle osseuse. Ce composé a diminué les niveaux de HDM2 et HDMX, et a augmenté les niveaux de p53 et p21. Globalement, la cytotoxicité induite par ce composé est médiatisée en partie via l'axe de signalisation HDM2-p21 et bien que p53 soit régulé à la hausse en réponse à ce traitement chimique, son activité cytotoxique n'est pas dépendante de p53. |
| Kinase Assay |
Ubiquitin Protease Assays
|
|
Les enzymes recombinantes dans 20 mM Tris-HCl (pH 8,0), 2 mM CaCl2 et 2 mM β-mercaptoéthanol sont incubées avec des plages de doses de P5091 pendant 30 min dans une plaque à 96 puits avant l'ajout d'Ub-PLA2 et de NBD C6-HPC ou d'Ub-EKL et de substrat EKL. La libération d'un produit fluorescent dans la plage linéaire de l'essai est surveillée à l'aide d'un lecteur de plaque à fluorescence Perkin Elmer Envision. Le véhicule (2% [v/v] DMSO) et 10 mM de N-éthylmaléimide (NEM) sont inclus comme contrôles.
|
|
| In vivo |
Dans les études de modèles tumoraux animaux, P5091 est bien toléré, inhibe la croissance tumorale et prolonge la survie. La combinaison de ce composé avec l'inhibiteur de HDAC SAHA déclenche une activité anti-MM synergique. |
Références |
|
Applications
| Méthodes | Biomarqueurs | Images | PMID |
|---|---|---|---|
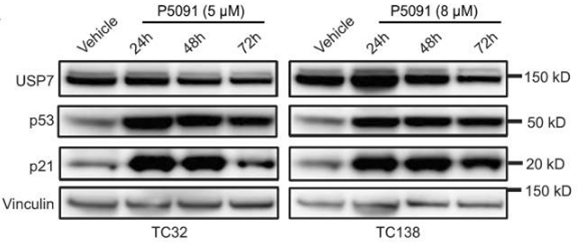
| Western blot | USP7 / p53 / p21 HAUSP / N-Myc |
|
30045945 |
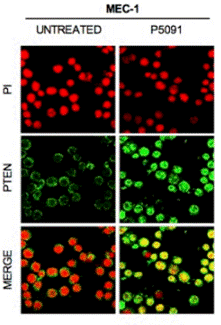
| Immunofluorescence | PTEN |
|
28418900 |
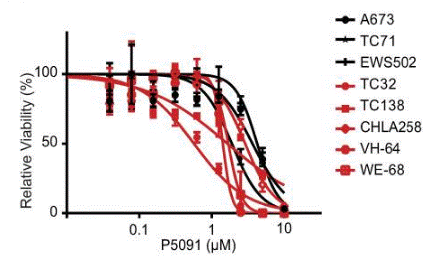
| Growth inhibition assay | Cell viability |
|
30045945 |
Support technique
Tel: +1-832-582-8158 Ext:3
Si vous avez dautres questions, veuillez laisser un message.
